siRNA는 왜 off-target effect에 취약한가?
RNAi기반의 유전자 발현조절 현상은 1998년 최초 발견되어, 2006년 노벨 생리의학상을 받기까지 10년이 채 걸리지 않을만큼 급속하게 연구가 진보되어 왔습니다.
잘 알려진 바와 같이, RNAi 현상은 이중 가닥의 RNA (dsRNA)가, Dicer라는 리보핵산 가수분해효소에 의해 잘려 생성된 21-23bp의 짧은 RNA조각을 통해 일어납니다. siRNA는 이 짧은 RNA조각과 유사하게 작용합니다. 세포내로 도입된 siRNA는 RISC (RNA induced silencing complex)복합체와 결합한 후 활성화된 Argonaute-2 에 의해 센스가닥이 분해됩니다. 활성화된 RISC 복합체와 결합된 안티센스 가닥은 표적하는 mRNA와 결합해 이것을 분해하여 최종적으로 단백질의 형성을 억제합니다.
1. 24-48시간 이내에 RNA loss를 볼 수 있고, 수일 내 단백질 발현이 억제되는 것을 확인할 수 있습니다.
2. siRNA는 약물처리와 같이 dose-dependent한 결과를 나타내며,
3. 일시적으로 발현되고, 처리량을 최소화하면 타겟 단백질 발현이 다시 회복됩니다.
4. Transfection을 통해 cell 에 직접 처리가 가능하여 적용이 간편합니다.
5. Delivery system이 적합한 경우, in vivo에도 손쉽게 적용할 수 있습니다.
이러한 잇점에도 불구하고 siRNA는 off-target effect라고 하는 극복해야 할 커다란 난관이 있습니다.
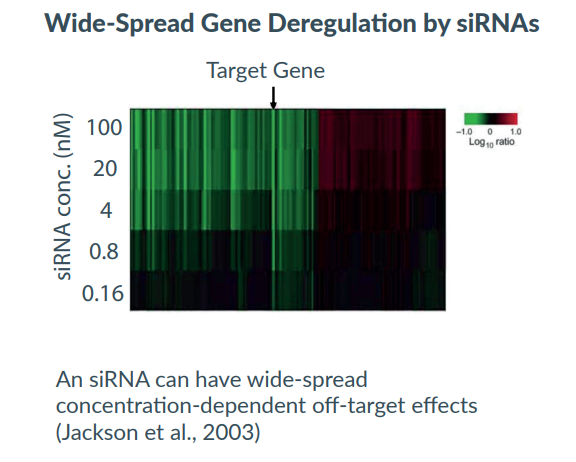
개개의 siRNA는 한 개의 타겟유전자를 발현억제 (down-regulation)하기 위해 디자인되었지만 아주 소량의 siRNA만으로도 타겟 유전자외 다른 유전자들이 감소되는 현상을 볼 수 있습니다. siRNA양을 증가시키면 이러한 off-target현상은 더욱 확대됩니다. 그림과 같이 Mapk14을 knockdown하기 위해 siRNA의 양을 증가시키면 mapk14외에 다른 유전자들의 발현도 함께 억제가 되는 현상이 관찰되는 것입니다.
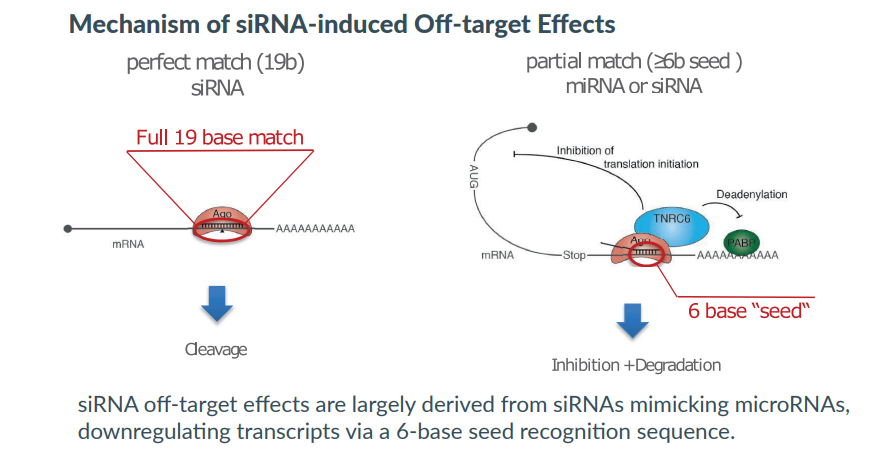
Off target effect의 주요 원인은 이 siRNA가 마치 내부에서 만들어진 microRNA처럼 작용하기 때문입니다.
siRNA는 애초에 타겟유전자에 perfect match 시퀀스로 디자인이 되었기 때문에 오직 타겟 유전자에만 결합하여 타겟 mRNA만 cleavage를 유도하는 것이 이상적일 것입니다. 그러나, 세포는 외부에서 들어온 siRNA를 miRNA와 잘 구분하지 못합니다.
microRNA는 5’의 몇 개의 base만 상보적이어도 결합이 되어 gene silencing을 유도합니다.
외부에서 넣어준 siRNA라도 siRNA의 5’ base 몇 개와 상보적인 시퀀스를 갖고 있는 전사체는 모두 off-target이 된다고 할 수 있습니다. 즉, seed 라고 불리는 2-7 nucleotides의 5’ base가 siRNA의 기능에서 매우 중요하다고 할 수 있습니다.
실제로 siRNA library를 이용한 실험에서 seed 시퀀스와 siRNA의 gene silencing효과의 관계를 분석해보면 상관관계가 매우 높다는 것을 확인할 수 있습니다.
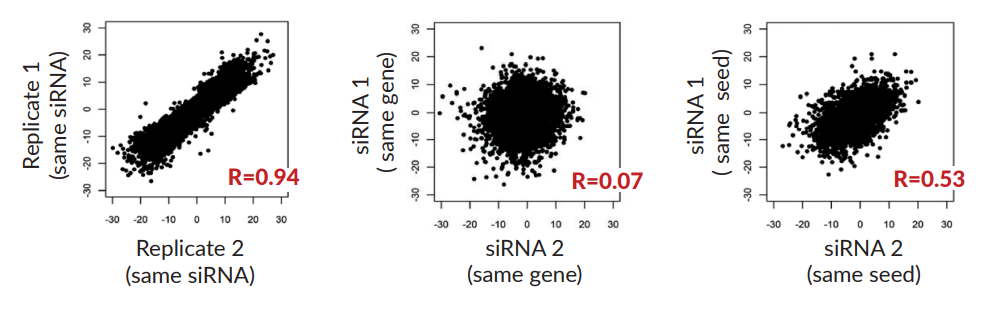
첫번째 data는 독립된 두 번의 반복한 RNAi screen결과를 비교 분석한 데이터입니다. 분석결과, 두 실험은 선형관계에 있는 것 나타나 RNAi screening의 기술적인 재현성 (duplicate)은 우수하다고 할 수 있습니다.
두번째 data는 타겟 유전자가 동일한 두 siRNA를 사용한 RNAi screen 결과의 비교 분석 데이터입니다. 그런데, 놀랍게도 두 RNAi screen은 상관관계가 거의 없는 것으로 나타났습니다.
세번째 data는 타겟유전자는 다르고, 6mer의 seed시퀀스가 동일한 두 siRNAs의 실험결과를 분석한 데이터입니다. 여기서 두 실험은 타겟유전자가 다름에도 불구하고 상관관계가 뚜렷한 것으로 나타났습니다. 즉, seed 서열이 RNAi결과에 영향을 미치는 보다 중요한 요소임을 알 수 있습니다.
siRNA의 이러한 off-target effect를 해결하기 위해 siTOOL사의 siPOOL siRNA pool 서비스를 제안합니다.